Da ich wohl nicht der einzige bin, der mit Bluthochdruck zu kämpfen hat, hier mal eine kleine Erklärung wie ich meine Blutdruckstatistiken mache. Zunächst mal braucht man natürlich ein Messgerät und sollte sich die Werte aufschreiben, ich notiere die Messergebnisse (eines am Morgen, eines am Abend) erstmal auf einem Zettel und einmal pro Woche wird das in den PC getippt.
Für die Erfassung nehme ich eine simple Textdatei die so aussieht:
|
1 2 3 4 5 |
Datum Zeit Sys Dia Pulse 14.01.10 22:00 165 96 58 15.01.10 05:30 150 97 47 15.01.10 22:45 160 107 55 16.01.10 07:50 145 105 45 |
Hier sieht man schon die Struktur der Datei. Wichtig ist die erste Zeile, denn sie enthält die Variablennamen welche später im R-Script verwendet werden. Die Datei hat dann pro Messwert eine Zeile in der das Datum (TT.MM.JJ) steht, danach die Uhrzeit und dann die drei Messwerte die das Blutdruckmessgerät ausspuckt.
Um diese Datei dann auswerten zu können braucht es das Statistik-Tool „R“ und ein paar Zeilen in R. Die Statistik nutzt zur graphischen Ausgabe die Bibliothek „ggplot2“ und bei Debian Wheezy ist leider eine etwas ältere Version dabei, die so nicht funktioniert. Darum habe ich nicht das Paket aus Wheezy genommen sondern es innerhalb von R mit
|
1 |
install.packages("ggplot2") |
nachinstalliert. Das in R geschriebene Programm fängt erst mal mit dem Einbinden der benötigten Bibliotheken an und definiert eine Funktion um die letzten „n“ Zeilen aus einem Dataframe zu extrahieren.
|
1 2 3 4 5 6 7 8 9 10 11 12 13 |
# Rework of the blood pressure stats # Author: Rainer Koenig library(ggplot2) # for the graphical output library(reshape) # for the melt function call # extract the last num lines from the dataset get_latest <- function(data, num) { rows <- nrow(data) cols <- ncol(data) start <- rows - num +1; return( data[start:rows,0:cols] ) } |
Das mit dem Begrenzen auf die letzten Zeilen kann man möglicherweise auch noch eleganter machen, aber es funktioniert auch so wie es dasteht. Nun folgt der Teil der die Datendatei einliest. Diese heißt bei mir sinnigerweise „daten.txt“ und das ist fest so im R-Code verankert.
|
14 15 16 17 18 |
# read the bloodpressure data from the file bp_data <- read.table("daten.txt", header=TRUE, sep=" ") bp_data$Timestamp <- strptime(paste(bp_data$Datum, bp_data$Zeit), "%d.%m.%y %H:%M") bp_data <- subset(bp_data, select=c(-Datum, -Zeit)) |
Die erste Zeile dieses Blocks liest die Daten ein. Datum und Uhrzeit wären jetzt als „characters“ hinterlegt was für die weitere Bearbeitung nicht so gut wäre. Daher wandelt die nächste Codezeile das in einen Zeitstempel um und die dritte Zeile verwirft die Spalten mit dem aus der Datei gelesenen Datum und der Zeit.
Für die nützlichen Graphiken betrachte ich nur den Zeitraum der letzten 60 Messungen.
|
19 20 21 22 23 24 |
# get a selection of the latest 60 measures bp_latest <- get_latest(bp_data, 60) # transform the data to a long table bp_plotdata <- melt(bp_latest, id="Timestamp") |
Der erste Block ruft die selbstdefinierte Funktion auf und gibt die letzten 60 Werte zurück. Der zweite Block transformiert die Daten so, dass wir einen Dataframe bekommen der den Zeitstempel enthält, eine Variable mit dem Spaltennamen (also „Sys“, „Dia“ und „Pulse“) sowie dem Value, also dem Wert der in der entsprechenden Spalte stand. All das macht die „melt“-Funktion recht problemlos. Der nächste Block produziert die Liniengraphik:
|
25 26 27 28 |
plot_latest_lines <- ggplot(bp_plotdata, aes(x=Timestamp, y=value, colour=variable)) + geom_line() + geom_point() + ylab("Messwert") + xlab("Datum") + scale_colour_discrete(name="Messwert") + scale_y_continuous(breaks=seq(40,200,by=10)) + labs(title="Bludruck (letzte 60 Werte)") |
Das Ergebnis dieser Zeile sieht dann so aus:
 Als nächstes habe ich spaßhalber mal mit einem Scatter-Plot experimentiert um zu sehen, ob es zwischen den „Sys“ und „Dia“ Werten eine Korrelation gibt. Also die Vermutung ist, dass ein hoher Wert vom einen auch einen hohen Wert vom anderen ergibt. Und die Punktewolke daher irgendwie „diagonal“ aussehen sollte.
Als nächstes habe ich spaßhalber mal mit einem Scatter-Plot experimentiert um zu sehen, ob es zwischen den „Sys“ und „Dia“ Werten eine Korrelation gibt. Also die Vermutung ist, dass ein hoher Wert vom einen auch einen hohen Wert vom anderen ergibt. Und die Punktewolke daher irgendwie „diagonal“ aussehen sollte.
Der Code hierfür sieht so aus:
|
29 30 31 |
plot_scatter_all <- ggplot(bp_data, aes(x=Sys, y=Dia)) + geom_point(alpha=0.2) + stat_smooth(method=lm) + labs(title="Korrelation Sys/Dia Werte") |
Und der damit erzeugte Scatterplot aus allen 2600 Messwerten die ich bis heute gesammelt habe sieht so aus und belegt die These: 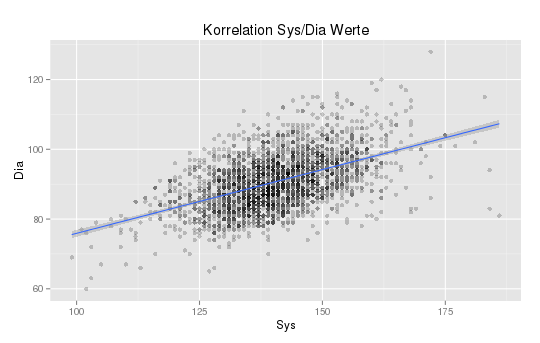 Viel interessanter als dieser Scatterplot den ich wie gesagt nur aus Experimentierfreude gemacht habe ist aber ein Histogramm der letzten Messwerte, also die Aussage in welchen Bereichen wie häufig Werte auftraten. Das Histogramm wird mit folgendem Code-Schnipsel erzeugt:
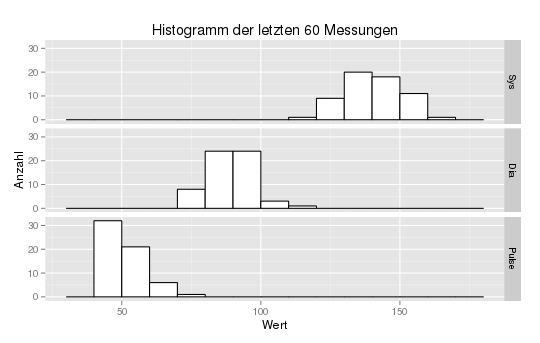
Viel interessanter als dieser Scatterplot den ich wie gesagt nur aus Experimentierfreude gemacht habe ist aber ein Histogramm der letzten Messwerte, also die Aussage in welchen Bereichen wie häufig Werte auftraten. Das Histogramm wird mit folgendem Code-Schnipsel erzeugt:
|
32 33 34 35 36 |
plot_histogram_latest <- ggplot(bp_plotdata, aes(x=value)) + geom_histogram(fill="white", colour="black", binwidth=10) + facet_grid(variable ~ .) + xlab("Wert") + ylab("Anzahl") + labs(title="Histogramm der letzten 60 Messungen") |
Und das damit erzeugte Histogram sieht dann so aus:
 Somit habe ich mit 36 Zeilen in „R“ eine recht aussagekräftige Blutdruckstatistik erzeugt die jederzeit mit aktuellen Daten ausgewertet werden kann. Damit ihr die Datei nicht aus Schnipseln zusammensetzen müsst könnt ihr sie hier auch runterladen. Ich habe sie mal mit der Endung „.txt“ versehen um sie hier in WordPress hochladen zu können (das mag nämlich keine „.R“-Dateien. Also ggf. noch anpassen.
Somit habe ich mit 36 Zeilen in „R“ eine recht aussagekräftige Blutdruckstatistik erzeugt die jederzeit mit aktuellen Daten ausgewertet werden kann. Damit ihr die Datei nicht aus Schnipseln zusammensetzen müsst könnt ihr sie hier auch runterladen. Ich habe sie mal mit der Endung „.txt“ versehen um sie hier in WordPress hochladen zu können (das mag nämlich keine „.R“-Dateien. Also ggf. noch anpassen.
[ratings]

Ich nutze R auch für die anzufertigen Statistiken. Fas einzige, was mir noch nie gelungen ist, ist das anfertigen einer Gaußschen Glockenkurve. Weißt du, wie man das macht?
Probier mal diesen Link.